AI人工智能 在Python中构建分类器
在本节中,我们将详细学习如何用Python构建各类分类器,涵盖朴素贝叶斯、支持向量机、逻辑回归、决策树和随机森林等常用模型,所有实现均基于Python 3和Scikit-learn库。
一、朴素贝叶斯分类器(Naive Bayes Classifier)
朴素贝叶斯是基于贝叶斯定理构建的分类技术,核心假设是“特征之间相互独立”——即某一类别中某个特征的存在,与其他特征的存在与否无关。
1. 构建所需条件
- 数据集:使用威斯康星州乳腺癌诊断数据库(Breast Cancer Wisconsin Diagnostic Database),包含569个肿瘤样本,每个样本有30个特征(如肿瘤半径、纹理、平滑度等),分类标签为“恶性(malignant)”或“良性(benign)”,可直接从Scikit-learn库导入。
- 模型选择:Scikit-learn提供三种朴素贝叶斯模型(GaussianNB、MultinomialNB、BernoulliNB),本文以高斯朴素贝叶斯(GaussianNB) 为例,适用于连续型特征。
2. 实现步骤
步骤1:导入库与数据集
## 导入Scikit-learn库
import sklearn
## 导入乳腺癌数据集
from sklearn.datasets import load_breast_cancer
## 加载数据集
data = load_breast_cancer()步骤2:规整数据
数据集为字典格式,提取核心信息(标签名、标签值、特征名、特征值)并创建变量:
## 分类标签名(恶性/良性)
label_names = data['target_names']
## 实际标签值(0=恶性,1=良性)
labels = data['target']
## 特征属性名(如平均半径、纹理等)
feature_names = data['feature_names']
## 特征属性值
features = data['data']步骤3:查看数据(可选,用于直观理解)
## 打印分类标签名
print(label_names) # 输出:['malignant' 'benign']
## 打印第一个样本的标签(0=恶性)
print(labels[0]) # 输出:0
## 打印第一个特征名
print(feature_names[0]) # 输出:mean radius(平均半径)
## 打印第一个样本的特征值
print(features[0]) # 输出:包含30个特征值的数组,第一个特征值为17.99(平均半径)从输出可知,第一个样本是平均半径为17.99的恶性肿瘤。
步骤4:划分训练集与测试集
使用train_test_split()函数将数据拆分,40%用于测试,60%用于训练:
from sklearn.model_selection import train_test_split
## 拆分数据(test_size=测试集占比,random_state=随机种子,保证结果可复现)
train, test, train_labels, test_labels = train_test_split(
features, labels, test_size=0.40, random_state=42
)步骤5:构建并训练模型
## 导入高斯朴素贝叶斯模块
from sklearn.naive_bayes import GaussianNB
## 初始化模型
gnb = GaussianNB()
## 拟合训练数据,完成模型训练
model = gnb.fit(train, train_labels)步骤6:模型预测与准确率评估
## 用测试集进行预测
preds = gnb.predict(test)
## 打印预测结果(0=恶性,1=良性)
print(preds)
## 输出:一串0和1的数组
[1 0 0 1 1 0 0 0 1 1 1 0 1 0 1 0 1 1 1 0 1 1 0 1 1 1 1 1 1
0 1 1 1 1 1 1 0 1 0 1 1 0 1 1 1 1 1 1 1 1 0 0 1 1 1 1 1 0
0 1 1 0 0 1 1 1 0 0 1 1 0 0 1 0 1 1 1 1 1 1 0 1 1 0 0 0 0
0 1 1 1 1 1 1 1 1 0 0 1 0 0 1 0 0 1 1 1 0 1 1 0 1 1 0 0 0
1 1 1 0 0 1 1 0 1 0 0 1 1 0 0 0 1 1 1 0 1 1 0 0 1 0 1 1 0
1 0 0 1 1 1 1 1 1 1 0 0 1 1 1 1 1 1 1 1 1 1 1 1 0 1 1 1 0
1 1 0 1 1 1 1 1 1 0 0 0 1 1 0 1 0 1 1 1 1 0 1 1 0 1 1 1 0
1 0 0 1 1 1 1 1 1 1 1 0 1 1 1 1 1 0 1 0 0 1 1 0 1]
## 导入准确率评估函数
from sklearn.metrics import accuracy_score
## 计算并打印准确率
print(accuracy_score(test_labels, preds))
## 输出:0.951754385965(约95.17%)结果说明
高斯朴素贝叶斯分类器在该数据集上的准确率达95.17%,能有效区分恶性和良性肿瘤。
二、支持向量机(SVM)分类器
支持向量机(SVM)是可用于分类和回归的监督学习算法,核心思想是:将每个数据点映射到n维空间(n=特征数),每个特征对应空间中的一个坐标,通过寻找最优超平面将数据划分为不同类别。
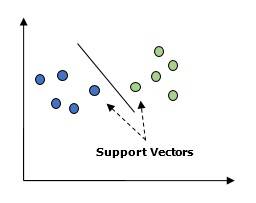
1. 构建所需条件
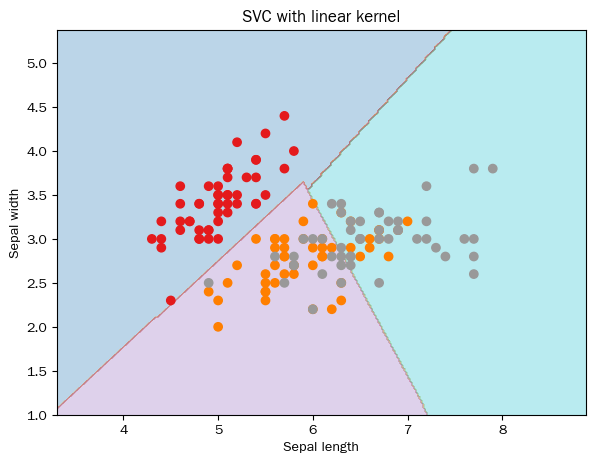
- 数据集:使用鸢尾花(Iris)数据集,包含3类鸢尾花(各50个样本),每个样本有4个特征(花萼长度、花萼宽度、花瓣长度、花瓣宽度)。
- 核函数:将低维空间映射到高维空间,解决线性不可分问题,本文使用线性核(linear),其他可选核函数包括多项式核(poly)、径向基核(rbf)、S型核(sigmoid)。
2. 实现步骤
步骤1:导入库与数据集
import pandas as pd
import numpy as np
from sklearn import svm, datasets
import matplotlib.pyplot as plt
## 加载鸢尾花数据集
iris = datasets.load_iris()步骤2:选择特征与标签
选取前两个特征(花萼长度、花萼宽度)用于可视化:
X = iris.data[:, :2] # 特征(前两个)
y = iris.target # 标签(3类鸢尾花)步骤3:创建可视化网格
为绘制分类边界,生成均匀分布的网格点:
## 定义网格的边界(特征值±1,留出余量)
x_min, x_max = X[:, 0].min() - 1, X[:, 0].max() + 1
y_min, y_max = X[:, 1].min() - 1, X[:, 1].max() + 1
## 定义网格步长
h = (x_max / x_min) / 100
## 生成网格点
xx, yy = np.meshgrid(np.arange(x_min, x_max, h), np.arange(y_min, y_max, h))
## 规整网格点格式,适配模型输入
X_plot = np.c_[xx.ravel(), yy.ravel()]步骤4:构建并训练SVM模型
## 正则化参数(C越大,对误分类的惩罚越重)
C = 1.0
步骤5:模型预测与可视化
## 创建SVM分类器对象(线性核,多分类采用ovr策略)
svc_classifier = svm.SVC(kernel='linear',
C=C, decision_function_shape='ovr').fit(X, y)
## 对网格点进行预测
Z = svc_classifier.predict(X_plot)
## 重塑预测结果格式,适配网格维度
Z = Z.reshape(xx.shape)
## 绘制分类边界与原始数据
plt.figure(figsize=(15, 5))
plt.subplot(121)
## 绘制分类边界(填充颜色)
plt.contourf(xx, yy, Z, cmap=plt.cm.tab10, alpha=0.3)
## 绘制原始数据点(按标签着色)
plt.scatter(X[:, 0], X[:, 1], c=y, cmap=plt.cm.Set1)
plt.xlabel('Sepal length(花萼长度)')
plt.ylabel('Sepal width(花萼宽度)')
plt.xlim(xx.min(), xx.max())
plt.title('SVC with linear kernel(线性核SVM分类器)')
plt.show()